

北京时间2020年8月10日晚23时,Nature Genetics在线发表了夏来新与肖姗团队合作的题为“N6-Methyladenosine co-transcriptionally directs the demethylation of histone H3K9me2”的研究成果。
研究首次发现m6A直接逆向调控抑制性组蛋白标记H3K9m2去甲基化的现象和机制,确立了RNA m6A修饰与组蛋白动态修饰之间的直接关系,揭示了环境和发育信号产生转录增强和记忆的新机制。
在该项研究中,为了筛选m6A共转录调控的表观修饰,夏来新团队建立了一个染色质修饰的报告系统,当报告基因转录本上发生了m6A修饰时,他们发现对应染色质上的抑制型组蛋白修饰标志物H3K9me2会被特异地去除,而其他的组蛋白修饰则变化不大。
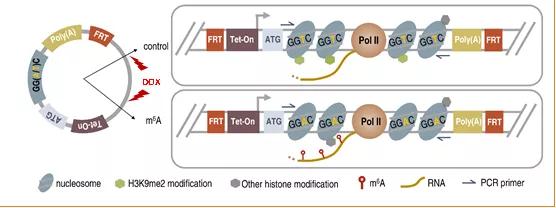
图1. 筛选转录过程中,响应m6A发生的表观修饰
他们继而在全基因组水平确认了m6A可以调控H3K9me2去甲基化,并发现m6A通过招募H3K9me2去甲基化酶KDM3B定位于染色质,进而控制对应区域的H3K9me2改变。
深入的分析表明m6A识别蛋白YTHDC1和KDM3B存在相互作用,且YTHDC1可以招募KDM3B到m6A相关的区域,从而促进对应染色质区域H3K9me2的去甲基化。
最后,研究者发现m6A能够促进H3K9me2调控的基因的转录。
综上所述,本研究揭示了RNA表观修饰信息通过共转录方式从RNA流动到染色质的新现象,建立了RNA修饰和组蛋白修饰之间相互作用的新机制,并为发育和表观记忆的研究提供了新视角,同时也为解答“RNA修饰在人类发育和相关疾病中的功能和调控”这一重大问题的研究提供了新线索。
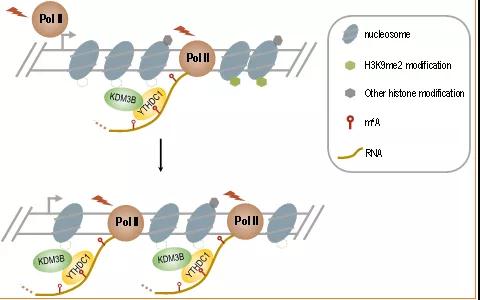
图2. m6A通过YTHDC1招募KDM3B调控H3K9me2去甲基化示意图
值得一提的是:共同第一作者排名第六位的李敏纯,是2015级基础医学专业的一名本科生。
相关论文信息:
DOI:10.1038/s41588-020-0677-3