

近日,南方医科大学基础医学院肿瘤研究所郝冰涛团队,在Nucleic Acids Research(IF:14.9, 1区)在线发表了题为“Three-way contact analysis characterizes the higher order organization of the Tcra locus”的研究论文。

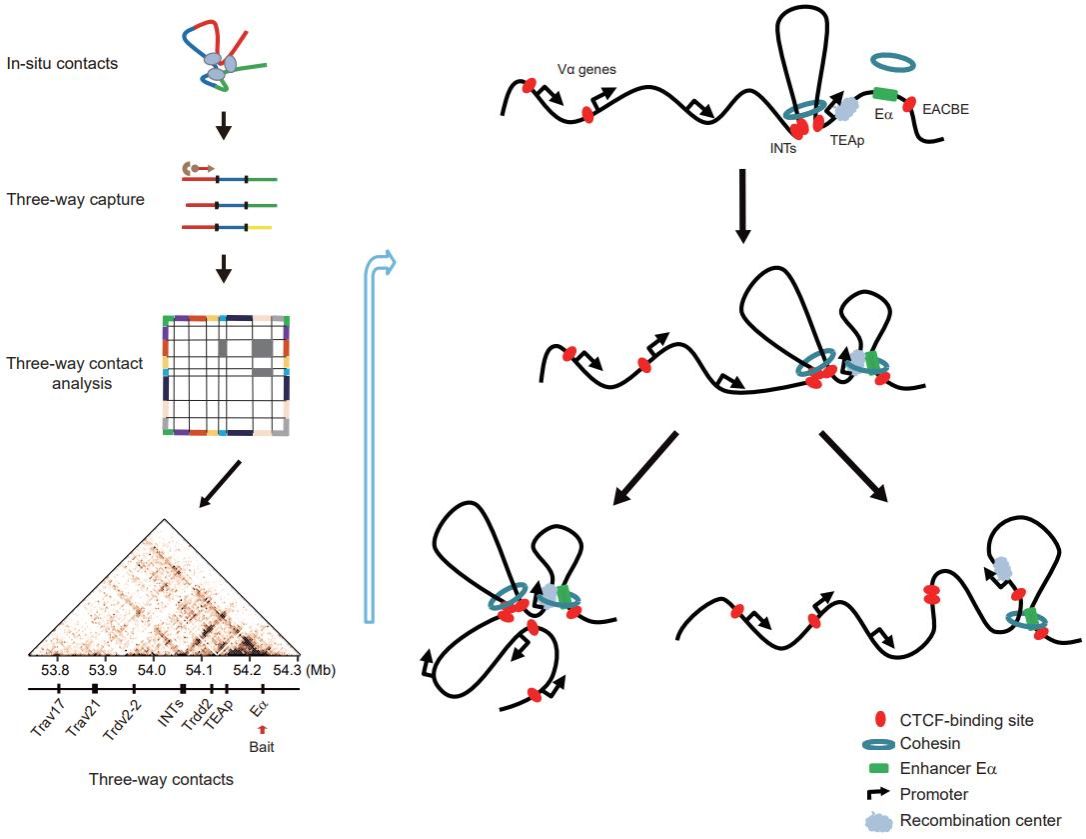
三维基因组学是以研究真核生物核内染色质空间构象及其对基因转录调控生物学效应为主要研究内容的一个新学科方向。现有基于高通量测序的三维基因组检测技术均以二阶为主,即一次只能检测两个基因组位点的空间互作。而高阶(即两个以上的基因组位点)空间互作尽管是核内染色质组织中更为常见现象,但是由于技术限制,高阶互作研究尚不充分。
T淋巴细胞和B淋巴细胞中高度多样化抗原受体的产生依赖于V(D)J重排。通常情况下,抗原受体基因的V片段远离D和J片段,因此需要在空间上靠近才能进行重排。Tcra-Tcrd基因座包含了编码T细胞受体TCRα和TCRδ基因。位于Tcra-Tcrd基因座下游的增强子Eα,通过激活Jα基因阵列上游启动子TEAp,在CD4+CD8+双阳性(DP)胸腺细胞中建立重排中心,在调节Tcra重排中发挥着至关重要的作用。Eα还通过长程相互作用调节染色质可及性和近端Vα基因的胚系转录。该团队之前也报道,位于Eα下游的CTCF结合位点EACBE增强了Eα与近端Vα基因之间的相互作用,从而促进了Tcra的初级重排。然而,Eα/EACBE物理介导Vα和Jα基因相互作用的直接证据仍然缺乏。
该研究团队利用基于3C的染色质相互作用技术:3C-HTGTS检测法,分析了Tcra基因座的高阶染色质结构。经分析表明,3C-HTGTS数据中存在足够的信息来检测多片段(多个基因组位点)相互作用。Tcra基因座的三片段相互作用分析表明,在CD4+CD8+双阳性胸腺细胞中,近端Jα基因、Vα基因和Eα/EACBE之间同时相互作用。随后,研究人员利用统计学方法Bait–SOI来识别高阶接触频率显著高于背景(存在“协同作用”)的基因组位点集,发现Eα/EACBE、近端Vα基因、近端Jα基因三个位点的相互作用概率显著高于仅涉及两个位点的相互作用概率,从而三者表现出协同模式。值得注意的是,INT2-TEAp形成的loop是一个突出的结构,它可能是使近端Jα基因和Vα基因空间上靠近的原因。此外,增强子Eα利用这一loop与近端Vα基因区域建立了物理上的空间靠近。这项研究深入揭示了Tcra基因座的高阶染色质结构,阐明了染色质的空间组织及其对V(D)J重排的影响。
南方医科大学基础医学院戴然然硕士(现在中山大学就读博士研究生)、郑州大学人民医院朱永常博士为本文共同第一作者,基础医学院郝冰涛教授、郑州大学廖世秀教授、南方医院刘楠教授为共同通讯作者。
全文链接:https://doi.org/10.1093/nar/gkad641